Flexibilität ist alles: Wie maschinelles Lernen beim Bau neuer Proteine hilft
Ein Forschungsteam des Heidelberger Instituts für Theoretische Studien (HITS) und des Max-Planck-Instituts für Polymerforschung (MPIP) hat ein Computermodell entwickelt, das lernt, Proteine mit sehr flexiblen Strukturen zu bauen – sogar mit Mustern, die in natürlichen Proteinen ungewöhnlich sind. Das Modell wurde jetzt auf der International Conference on Machine Learning (ICML) präsentiert. Es markiert einen Schritt hin zum Ziel, neue Proteine für Anwendungen in der Biotechnologie, in Therapien und in der Umweltforschung zu konstruieren.
Proteine kommen in der Natur in einer großen Vielfalt vor, und als molekulare Werkzeuge erfüllen sie unterschiedlichste Aufgaben im Organismus. Dennoch kann die Entwicklung neuer, in der Natur nicht beobachteter Proteine zu neuen Funktionen führen und Probleme in Medizin oder Materialwissenschaft lösen. Gerade in den letzten Jahren begann ein „goldenes Zeitalter“ des De-novo-Designs von Proteinen: Methoden des maschinellen Lernens ermöglichten eine bislang nicht dagewesene Genauigkeit der Modellierung. Dadurch können Forschende Proteinstrukturen mit spezifischen funktionellen Eigenschaften entwerfen, die nie zuvor beobachtet wurden. Dies ist besonders interessant für biotechnologische Anwendungen, die Entwicklung von Therapien und Probleme im Bereich der Nachhaltigkeit, Nachhaltigkeitsprobleme, wie zum Beispiel den Abbau von Plastik.
Eines der wichtigsten Merkmale funktioneller Proteine – große Biomoleküle mit komplexen Strukturen – ist ihre strukturelle Flexibilität: Sie wackeln, zappeln und verändern ihre Form. Beim derzeitigen Protein-Design fehlt dieses wichtige Merkmal weitgehend. Für ein Forschungsteam des Heidelberger Instituts für Theoretische Studien (HITS) und des Max-Planck-Instituts für Polymerforschung (MPIP) war dies der Ausgangspunkt, sich mit der Frage zu befassen, ob man Proteine mit einer maßgeschneiderten Flexibilität von Grund auf entwerfen kann. Die Ergebnisse ihrer Arbeit präsentierten sie jetzt auf der International Conference on Machine Learning (ICML) in Vancouver, Kanada.
Schnell und effizient: Ein System aus zwei Bausteinen
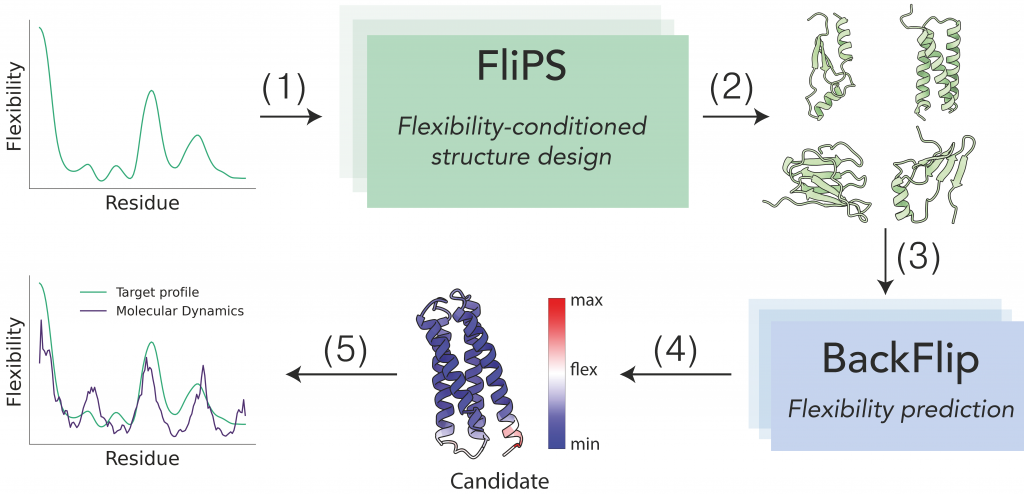
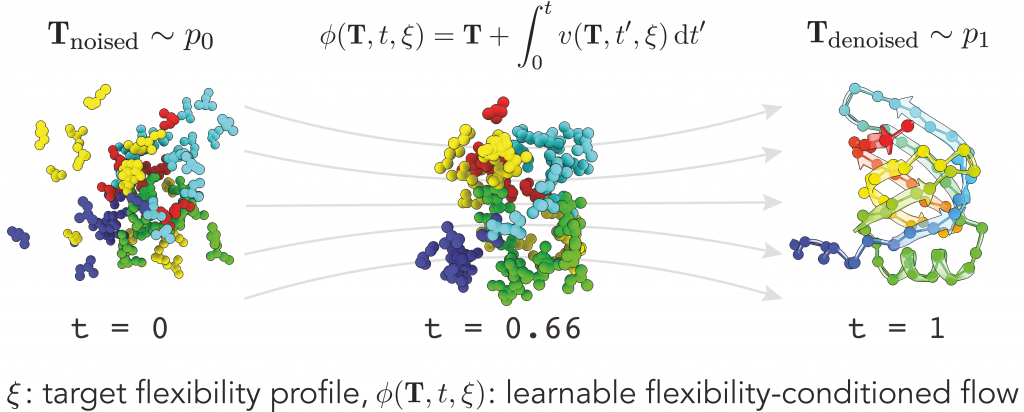
„Wir wollten ein Modell entwickeln, das lernt, Proteine so zu erzeugen, dass ihre Strukturen an einer bestimmten Stelle in einem bestimmten Maße flexibel sind“, so Erstautor Vsevolod Viliuga (MPIP). Deshalb erarbeiteten sie ein System, in dem flexible Proteinstrukturen generiert werden können. Dieses System besteht aus zwei Bausteinen: Erstens einem neuronalen Netzwerk, darauf trainiert, die Flexibilität von sogenannten „Backbones“ vorherzusagen – eine Reihe von Atomen, die als gefaltete Kette das „Rückgrat“ der Makromoleküle bilden. Und zweitens einem generativen Modell für Proteinstrukturen.
“Natürliche Proteine erfüllen ihre Aufgaben so hervorragend, weil sie überall, wo sie gebraucht werden, flexibel einsetzbar sind“, sagt Ko-Autor Leif Seute (HITS). „Wir können jetzt neue Proteine erzeugen, die diese Schlüsseleigenschaft nachahmen.“ HITS-Gruppenleiter Jan Stühmer ergänzt: „Das ist eine Erweiterung des „Geometric Algebra Flow Matching“, kurz: GAFL, das wir letztes Jahr entwickelt haben.“ GAFL ist dreimal schneller als vergleichbare Modelle und erreicht nicht nur hohe Werte in der Designfähigkeit – die Ergebnisse sind ebenfalls natürlichen Proteinen in verschiedenen Kriterien ähnlicher.
Das Team konnte zeigen, dass das Modell Proteine mit den gewünschten Flexibilitätsmustern erzeugen kann, sogar für Muster, die in natürlichen Proteinen ungewöhnlich sind. Frauke Gräter, Teamleiterin auf Seiten des MPIP, stellt zusammenfassend fest: „Diese Arbeit ist ein Schritt in Richtung des anspruchsvollen Ziels, neue Proteine für Anwendungen zu entwerfen, bei denen Flexibilität erforderlich ist, wie beispielsweise bei Katalysatoren wie Enzymen.“
Publikation:
Vsevolod Viliuga,Leif Seute, Nicolas Wolf, Simon Wagner, Arne Elofsson, Jan Stühmer, Frauke Gräter: Flexibility-conditioned protein structure design with flow matching.
https://icml.cc/virtual/2025/poster/46289
Die Arbeit wurde von der Klaus Tschira Stiftung (HITS Lab) gefördert.
Wissenschaftlicher Kontakt:
Jun.-Prof. Dr. Jan Stühmer
Junior Group Leader
Machine Learning and Artificial Intelligence
Heidelberger Institut für Theoretische Studien (HITS)
https://www.h-its.org/de/people/dr-jan-stuhmer/
Prof. Dr. Frauke Gräter
Director, Abteilungsleiterin “Biomolekulare Mechanik”
Max-Planck-Institut für Polymerforschung (MPIP)
https://www.mpip-mainz.mpg.de/1001466/01_Direktor
Medienkontakt:
Dr. Peter Saueressig
Head of Communications
Heidelberger Institut für Theoretische Studien (HITS)
+49 (0)6221 533 245
peter.saueressig@h-its.org
Teresa Petry
Presse- und Öffentlichkeitsarbeit
Max-Planck-Institut für Polymerforschung (MPIP)
+49 (0)6131 379-119
pr@mpip-mainz.mpg.de
Über das HITS
Das HITS (Heidelberger Institut für Theoretische Studien) wurde 2010 von dem Physiker und SAP-Mitbegründer Klaus Tschira (1940-2015) und der Klaus Tschira Stiftung als privates, gemeinnütziges Forschungsinstitut gegründet. Es betreibt Grundlagenforschung in den Naturwissenschaften, der Mathematik und der Informatik. Zu den Hauptforschungsrichtungen zählen komplexe Simulationen auf verschiedenen Skalen, Datenwissenschaft und -analyse sowie die Entwicklung rechnergestützter Tools für die Forschung. Die Anwendungsfelder reichen von der Molekularbiologie bis zur Astrophysik. Ein wesentliches Merkmal des Instituts ist die Interdisziplinarität, die in zahlreichen gruppen- und disziplinübergreifenden Projekten umgesetzt wird. Die Grundfinanzierung des HITS wird von der Klaus Tschira Stiftung bereitgestellt.