τRAMD – Berechnung von Protein-Ligand-Dissoziationsraten und Erforschung von Dissoziationsmechanismen
Forscher/-innen der Molecular and Cellular Modeling (MCM) Gruppe am HITS haben τRAMD (τ-Random Acceleration Molecular Dynamics) entwickelt: ein effizienter Workflow, der die Vorhersage relativer Wirkstoff-Protein-Verweilzeiten und die Analyse von Dissoziationsmechanismen auf automatisierte Weise ermöglicht. Als solches ist τRAMD ein flexibles Werkzeug, das in Arbeitsabläufe des rationalen Wirkstoffdesigns zum Entwurf neuer Moleküle oder zur Ligandenoptimierung implementiert werden kann.
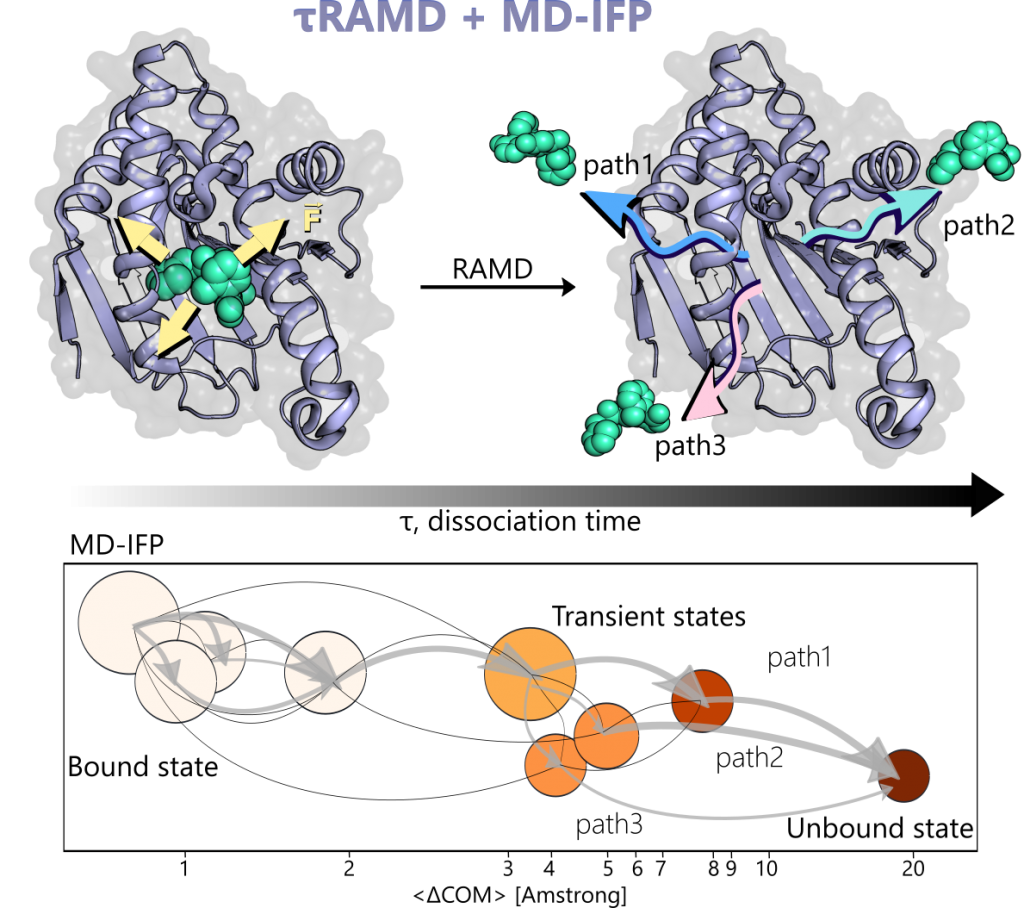
Das τRAMD-Protokoll wendet RAMD an, um den Austritt eines kleinen Moleküls aus einem Zielrezeptor durch die Anwendung einer adaptiven, zufällig orientierten Kraft auf den Liganden zu beschleunigen. Der Workflow ermöglicht auch die Untersuchung von Dissoziationsmechanismen und die Charakterisierung von Übergangszuständen durch das MD-IFP (Molecular Dynamics – Interaction Fingerprint) Post-Analyse-Tool.
Zusätzlich zu den Implementierungen in den Softwarepaketen NAMD und AMBER wurde τRAMD auch in die frei verfügbare molekulare Simulationsmaschine GROMACS für Simulationen auf CPU- oder GPU-Knoten implementiert.
Weitere Quellen:
Kokh DB, Doser B, Richter S, Ormersbach F, Cheng X, Wade RC (2020). A workflow for exploring ligand dissociation from a macromolecule: Efficient random acceleration molecular dynamics simulation and interaction fingerprint analysis of ligand trajectories, J. Chem. Phys. 153(12):125102
Kokh DB, Amaral M, Bomke J, Grädler U, Musil D, Buchstaller H, Dreyer MK, Frech M, Lowinski M, VKokh DB, Amaral M, Bomke J, Grädler U, Musil D, Buchstaller H, Dreyer MK, Frech M, Lowinski M, Vallee F, Bianciotto M, Rak A, Wade RC (2018). Estimation of Drug-Target Residence Times by τ-Random Acceleration Molecular Dynamics Simulations, J. Chem. Theory Comput. 14(7):3859-3869
Über das HITS
Das HITS (Heidelberger Institut für Theoretische Studien) wurde 2010 von dem Physiker und SAP-Mitbegründer Klaus Tschira (1940-2015) und der Klaus Tschira Stiftung als privates, gemeinnütziges Forschungsinstitut gegründet. Es betreibt Grundlagenforschung in den Naturwissenschaften, der Mathematik und der Informatik. Zu den Hauptforschungsrichtungen zählen komplexe Simulationen auf verschiedenen Skalen, Datenwissenschaft und -analyse sowie die Entwicklung rechnergestützter Tools für die Forschung. Die Anwendungsfelder reichen von der Molekularbiologie bis zur Astrophysik. Ein wesentliches Merkmal des Instituts ist die Interdisziplinarität, die in zahlreichen gruppen- und disziplinübergreifenden Projekten umgesetzt wird. Die Grundfinanzierung des HITS wird von der Klaus Tschira Stiftung bereitgestellt.